Sintesi proteica

La biosintesi proteica (PBS) è la formazione di nuove proteine nelle cellule . In questo processo, centrale per tutti gli esseri viventi, si costruisce una proteina a partire da amminoacidi sulla base delle informazioni genetiche .
La sintesi di una proteina dai suoi elementi costitutivi, gli amminoacidi proteinogenici , avviene nel contesto dell'espressione genica sui ribosomi . La sintesi proteica ribosomiale viene anche chiamata traduzione , poiché la sequenza di basi di un RNA messaggero (mRNA) viene tradotta nella sequenza di amminoacidi di un peptide . Questo viene fatto assegnando continuamente il corrispondente anticodone di un RNA di trasferimento (tRNA) a ciascun codone dell'mRNA e legando il suo amminoacido trasportato individualmente a quello vicino ( legame peptidico ), in modo da creare una catena con una sequenza amminoacidica caratteristica . Questo polipeptide può dispiegarsi nel mezzo circostante per formare una struttura strutturata di forma tridimensionale , la proteina nativa . Viene spesso modificato in seguito, modificato post-traduzionalmente attraverso scissioni, conversioni e addizioni .
Mentre nelle cellule procariotiche ( prociti ) il DNA circolare è libero nel citosol e la sintesi proteica ribosomiale avviene per lo più immediatamente e prontamente con l'mRNA appena creato, la situazione nelle cellule eucariotiche ( euciti ) è più complicata. Per il genoma, che viene distribuita su diversi cromosomi , il nucleo cellulare (nucleo) è un separato vano , nel caryoplasm di cui trascrizione anche avviene. La copia di RNA principalmente estratta (hnRNA) viene prima stabilizzata, rivista e preparata per l'esportazione nucleare prima che passi attraverso un poro nucleare come mRNA e raggiunga il citoplasma , che contiene le subunità dei ribosomi. Questa divisione spaziale e il percorso del processo in più fasi consentono quindi ulteriori modi per modificare un (hn) RNA post-trascrizionale e per regolare l'espressione genica o per escludere determinati modelli di RNA dalla biosintesi proteica ( arresto del gene ).
Alcuni tipi di batteri , archeobatteri e funghi possono costruire proteine speciali rispetto proteina ribosomale sintesi multi-enzima complessi un nonribosomal peptide sintesi consentire (NRPS).
trascrizione
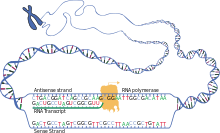
Nella prima fase della biosintesi proteica in una cellula, vengono ricercate, lette e trascritte sezioni di geni sul DNA a doppio filamento in molecole di RNA a filamento singolo . Durante questo processo, alla sequenza delle basi azotate del DNA ( adenina , guanina , citosina , timina ) vengono assegnate in modo complementare le basi azotate dei mattoni dell'RNA ( uracile , citosina, guanina, adenina). Il ribosio al posto del desossiribosio e l'uracile al posto della timina compaiono nel trascritto dell'RNA che viene poi legato per formare un filamento . L'informazione genetica è contenuta nella sequenza di basi, un codogeno sul DNA è trascritto in un codone sull'acido ribonucleico messaggero, o mRNA in breve .
Per la trascrizione di un gene oltre a diversi altri fattori è necessaria una RNA polimerasi come enzima la cui struttura continua del polimero di RNA dipende dallo stampo di DNA catalizzato . I ribonucleosidi trifosfati di appaiamento delle basi ( UTP , CTP , GTP e ATP ) assegnati allo stampo come elementi costitutivi sono collegati tra loro per formare un polinucleotide di un RNA, ciascuno con la scissione di due gruppi fosfato dei trifosfati . Possono esistere diversi tipi di RNA polimerasi per la trascrizione di geni che codificano per proteine mediante un mRNA, e per quelle di altri geni, ad esempio per la formazione di un rRNA o di un tRNA .
Negli eucarioti la trascrizione avviene nel nucleo ; quindi, l'mRNA deve essere esportato dal nucleo al citosol , poiché lì avviene la traduzione. I procarioti, d'altra parte, non hanno un compartimento centrale; la trascrizione avviene qui insieme alla traduzione nel plasma cellulare.
Modifica post-trascrizionale
- giuntura
- Nel caso degli eucarioti, dopo la trascrizione pura, gli introni non codificanti contenuti nel pre-mRNA risultante devono essere tagliati in modo che rimangano solo gli esoni richiesti . Questo processo è chiamato splicing . Le sequenze di consenso vengono utilizzate per identificare gli introni. Durante lo splicing, diversi snRNP si legano nell'area degli introni e nelle giunzioni esone-introne. Con la formazione dello spliceosoma, questi portano alla scissione dei legami fosfodiestere e quindi al taglio degli introni. Allo stesso tempo gli esoni sono legati. Lo splicing avviene anche con rRNA e tRNA.
- tappatura
- Durante questo periodo avviene anche il cosiddetto capping , durante il quale viene aumentata la stabilità dell'RNA. Viene attaccata una cosiddetta struttura 5'-cap , con l'estremità 5' del pre-mRNA sintetizzato che viene convertita in una struttura chiamata "cap" e l'mRNA prima della digestione da parte delle 5'-esonucleasi e delle fosfatasi protegge.
- poliadenilazione
- Durante la poliadenilazione , le code di poli (A) sono attaccate all'estremità 3' appena creata dell'RNA ( lunghezza fino a 250 nucleotidi ). Questa coda poli (A) facilita l'esportazione dell'mRNA nel citoplasma e protegge anche l'estremità 3' dalla degradazione enzimatica.
- edizione RNA
- Con l' edizione RNA , singole o più basi azotate della molecola di RNA vengono cambiate (modificate), inserite (inserite) o tagliate (cancellate) dopo la trascrizione. Ad esempio, l'editing può comportare un nuovo codone di stop sull'mRNA che è a monte del precedente; la traduzione poi si interrompe qui e si forma l'isoforma più corta di una proteina. L'editing dell'RNA si verifica solo in alcuni organismi, cellule o organelli cellulari ed è spesso limitato a particolari sequenze nucleotidiche.
Traduzione
Under Translation si riferisce alla traduzione della sequenza di basi dell'mRNA nella sequenza amminoacidica della proteina al ribosoma . Nell'mRNA, tre basi consecutive, una tripletta di basi , formano un codone all'interno della cornice di lettura aperta che codifica per un amminoacido (vedi codice genetico ). Sul ribosoma i codoni vengono tradotti in amminoacidi secondo la loro sequenza e questi vengono sequenzialmente legati a formare un polipeptide .
Per formare un legame peptidico tra due amminoacidi, devono essere portati in prossimità spaziale l'uno con l'altro. Ciò richiede la superficie di una grande struttura supramolecolare. Questo compito è svolto dai ribosomi, composti da una subunità piccola e una grande, che formano due siti di legame adiacenti: il sito A e il sito P.
Poiché non esiste una relazione strutturale tra un codone e l'aminoacido associato, è necessario un pezzo intermedio che, da un lato, leghi l'amminoacido e, dall'altro, riconosca il codone associato sull'mRNA. Questa funzione di mediazione è svolta dal trasferimento di molecole di acido ribonucleico , vari tRNA , come “trasportatori” di amminoacidi con regioni di riconoscimento. Hanno due siti di legame esposti, il sito di legame degli amminoacidi e l' anticodone . I siti di legame degli amminoacidi dei tRNA sono specificamente caricati con l'amminoacido appropriato dalle amminoacil-tRNA sintetasi . Il tRNA riconosce il codone complementare sull'mRNA con l'anticodone e si lega specificamente ad esso.
Il processo di traduzione in quanto tale può essere suddiviso in tre fasi: la fase iniziale, la fase di allungamento e infine la conclusione:
- Fase iniziale
- Se un mRNA precedentemente sintetizzato raggiunge un ribosoma , la piccola subunità del ribosoma migra lungo l'mRNA fino a incontrare il codone di inizio AUG. Il corrispondente tRNA della metionina con l' anticodone UAC è attaccato al codone ( complesso di inizio ).
- Fase di allungamento
- Con la scissione del GTP , ora si accumula anche la grande subunità del ribosoma e inizia l'allungamento.
- Durante la fase di iniziazione, il tRNA della metionina si trova sul sito di legame P, in modo che il successivo tRNA possa attaccarsi al sito di legame A. Una peptidil transferasi lega la metionina del primo tRNA con l'aminoacido del successivo tRNA; questa formazione di un dipeptide avviene nel sito di legame A. Infine, le unità ribosomiali migrano una tripletta di basi.
- Il tRNA con il dipeptide si trova ora nel sito di legame P da cui ha spostato il primo tRNA, ora non caricato, e il successivo tRNA, il cui anticodone è complementare alla tripletta di basi del filamento di mRNA, può nuovamente legarsi al filamento libero A-binding sito si adatta.
- Cessazione
- Se un ribosoma che si muove lungo l' mRNA incontra uno dei tre codoni di stop , la traduzione inizialmente si ferma, poiché non ci sono molecole di tRNA corrispondenti che sono codificate per un amminoacido (soppressione). Sono sostituiti dai cosiddetti fattori di terminazione o rilascio (RF), che si legano al sito A e modificano la specificità del substrato della peptidil transferasi in modo tale da attivare una molecola d'acqua invece di un tRNA AA. Il suo attacco nucleofilo al legame tra la catena peptidica e il tRNA rilascia infine la proteina sintetizzata e separa l'mRNA dal ribosoma.
Modifica co- e post-traduzionale
Le catene polipeptidiche di alcune proteine sono già modificate durante la traduzione (cotraduzionale) da speciali enzimi, ma nella maggior parte dei casi le proteine vengono modificate solo dopo che la traduzione è stata completata (post-traduzionale). Mentre gli chaperon influenzano il processo di formazione del ripiegamento proteico, da cui dipende anche l'associazione a complessi proteici, una cellula ha anche una moltitudine di possibilità per modificare in modo specifico la struttura di una proteina, per creare in questo modo specie proteiche funzionalmente diverse e quindi per espandere il proteoma attraverso modificazioni .
Queste modifiche includono la scissione di singoli amminoacidi terminali o le sequenze peptidiche più lunghe nel caso di proteine precursori , l'introduzione di legami aggiuntivi, ad es. B. ponti disolfuro tra residui di cisteina , o gruppi funzionali, come idrossilazioni di amminoacidi (da prolina a 4- idrossiprolina da parte della prolil-4-idrossilasi , da lisina a idrossilisina da parte della lisilidrossilasi ), nonché ossidazioni (es. reticolazione mediante residui di lisina da parte della lisil ossidasi ), carbossilazione o decarbossilazione e numerose altre. Ad esempio, le glicoproteine si formano per glicosilazione e le lipoproteine per acilazione e prenilazione .
Le singole fasi di modifica sono catalizzate ciascuna da enzimi speciali, la cui presenza è spesso limitata a determinati organelli, cellule o tessuti. Inoltre, la sequenza delle fasi di modifica o il loro andamento temporale può essere variata, a seconda dell'ambiente cellulare, della fase di sviluppo o delle condizioni ambientali. La molecola del collagene, ad esempio, subisce una serie di modificazioni post-traduzionali, alcune delle quali avvengono solo nello spazio extracellulare.
Targeting e trasporto delle proteine
Poiché molte proteine come destinazione ( target inglese ) non è il citosol, ma lo spazio extracellulare , la membrana cellulare , gli organelli come i cloroplasti , i mitocondri , i perossisomi , il nucleo o il reticolo endoplasmatico hanno la cellula ha diversi meccanismi per trascorrere lì le proteine. Queste proteine di solito contengono una sequenza segnale N- o C-terminale , che può essere strutturata in modo molto diverso a seconda del meccanismo bersaglio. In alcuni casi non esiste una sequenza segnale terminale, ma segnali interni della catena peptidica, che determinano la posizione bersaglio della proteina.
- Le proteine che prendono di mira il reticolo endoplasmatico (ER) portano una specifica sequenza N-terminale che è riconosciuta da un complesso proteina-RNA, la Signal Recognition Particle (SRP). Il complesso SRP-peptide-ribosoma viene quindi reclutato nel reticolo endoplasmatico, dove viene riconosciuto e legato. La traduzione continua attraverso la membrana. I ribosomi attaccati danno l'impressione di un "pronto soccorso ruvido". Vedi trasporto proteico cotraduzionale . Il controllo di qualità della proteina appena sintetizzata avviene nel reticolo endoplasmatico .
- Le proteine che devono essere introdotte nei cloroplasti hanno una sequenza segnale N-terminale che di solito è fosforilata precocemente . Le proteine Hsp70 , 14-3-3 e Toc64 possono anche svolgere un ruolo nel riconoscimento e nella trasmissione attraverso l'interazione con il precursore proteico. Dopo essere arrivato sulla superficie del cloroplasto, il complesso proteina-precursore viene riconosciuto dalle strutture recettoriali dell'apparato translocone della membrana esterna dei cloroplasti ( Translocon Of Outer Chloroplast Membrane , TOC). Sotto GTP - l' idrolisi della proteina viene quindi importata nello spazio intermembrana, o direttamente dal Translokonapparat ( TIC ) della membrana interna del cloroplasto nello stroma importato. Almeno 4 vie sono utilizzate per l'importazione nella membrana o nel lume dei tilacoidi , che sono indicati come Sec-dipendenti , SRP-dipendenti , delta-pH/Tat-dipendenti o spontanei .
- Finora sono state descritte tre diverse vie di importazione per il mitocondrio di lievito e cellule animali:
- La via di importazione della presequenza le cui proteine trasportano un'alfa elica anfifila N- terminale. Queste proteine sono principalmente destinate alla matrice, alla membrana interna o allo spazio intermembrana.
- La via di importazione delle proteine di trasporto per le proteine della membrana interna, che trasportano vari segnali interni.
- Il percorso di importazione delle proteine della membrana dell'involucro esterno, che viene utilizzato per integrare le proteine con un motivo beta-barile . Anche qui ci sono segnali all'interno della sequenza.
- Tutte e tre le vie di importazione iniziano dall'apparato di traslocazione mitocondriale nella membrana esterna ( TOM ), che ha diversi recettori. I recettori Tom20 e Tom22 riconoscono il segnale N-terminale e trasmettono la proteina precursore al poro Tom40 . Il recettore Tom70 riconosce i segnali interni delle proteine destinate alla membrana esterna.
- Dopo l'importazione nello spazio intermembrana, i percorsi si separano: le proteine con il motivo a botte beta, destinate alla membrana esterna, vengono integrate nella membrana dal complesso SAM (macchine di smistamento e assemblaggio). Le proteine delle altre due vie di importazione sono dirette a diversi complessi TIM : le proteine con una presequenza sono riconosciute dal complesso TIM23, mentre le proteine per la membrana interna sono riconosciute dal complesso TIM22 .
- La pre-sequenza è ad opera dell'enzima MPP (in inglese mitochondrial processing peptidase away).
Oltre alle sequenze segnale descritte sopra, la glicosilazione consente il targeting per l'incorporazione nella membrana cellulare o per l' esocitosi . Entrambi i percorsi di solito conducono attraverso le vescicole del Golgi .
Guarda anche
link internet
Evidenze individuali
- ↑ Matthias Strieker, Alan Tanović, Mohamed A. Marahiel: sintetasi peptidiche non ribosomiali : strutture e dinamiche. In: Opinione corrente in biologia strutturale. Volume 20, numero 2, aprile 2010, pp. 234-240, doi : 10.1016 / j.sbi.2010.01.09 , PMID 20153164 .
- ↑ Gavin J. Williams: Ingegneria sintasi polichetidica e sintetasi peptidica non ribosomiale. In: Opinione corrente in biologia strutturale. Volume 23, numero 4, agosto 2013, pp. 603-612, doi : 10.1016 / j.sbi.2013.06.012 , PMID 23838175 .
- ↑ a b c Traduzione . ( Spektrum.de [consultato il 15 giugno 2018]).